近日,电子科学与技术学院林雁勤教授团队在基于深度学习的核磁共振(NMR)谱图非均匀采样(NUS)重建方面取得重要突破,相关成果以“Deep learning network for NMR spectra reconstruction in time-frequency domain and quality assessment”为题,发表在Nature Communications期刊上。这一成果将会帮助科学家快速获取高质量的NMR谱图,并评估其可靠性,助力蛋白质结构解析和作用机制阐述,为生物医学研究和药物研发提供技术支持,助推健康中国建设。
多维核磁共振谱的获取通常耗时较长,非均匀采样(NUS)技术通过减少间接维采样点数显著缩短采样时间,但需要高效的重建算法来获得高质量谱图。传统重建算法虽有效,但存在参数调整复杂等问题。现有的深度学习(DL)方法局限于单域(时域或频域)重建,存在丢峰和伪峰等问题。此外,该领域存在一个长期未解决的重要问题,即如果没有全采样谱图,用户就无法评估重建谱的质量,困扰了实际应用。
该团队提出联合时频域的深度学习重建网络JTF-Net,如图1(a)所示。该网络在重建过程中结合了NMR谱的时频域信息,与传统算法和单域DL算法相比实现了更优的重建效果。此外,JTF-Net在重建过程中通过多次重建,不仅能得到重建谱图,还能获取偶然不确定性和感知不确定性,如图1(b)所示。JTF-Net通过对大量模拟谱图的重建,建立了质量空间。基于此质量空间,在实际使用时获取REQUIRER(NMR谱图重建领域首个无参考质量评估指标,Reconstruction quality assurance ratio)来评估重建谱图的质量。
在与传统重建算法和单域深度学习算法的比较中,JTF-Net在不同样品和不同类型谱图上均实现了最优效果。其中,JTF-Net在T4L L99A蛋白不同采样率的1H-15N HSQC谱的重建中实现了最低的RLNE指标,如图2所示。此外,JTF-Net也重建了BMRB数据库中13中不同蛋白质的不同类型的NMR谱图,也同样实现了最优效果。
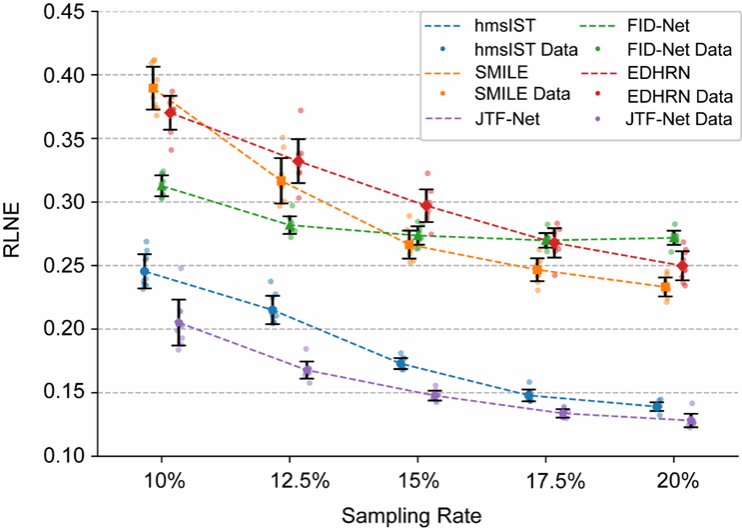
图2. 在不同采样率(10%、12.5%、15%、17.5% 和 20%)下,JTF-Net、hmsIST、SMILE、FID-Net 和 EDHRN 之间的 RLNE 比较。
REQUIRER也同样被验证是有效的。在GB1蛋白(图3)、T4L L99A蛋白和天青蛋白的验证中,REQUIRER根据重建谱图的不确定性,在不需要全采样谱图的情况下准确反映出了重建谱图的质量,可在实际非均匀采样场景下使用。

图3. JTF-Net 重建的具有不同信噪比 (SNR) 的GB1蛋白的2D 15N-1H HSQC谱。图中给出了它们的 REQUIRER指标。
该工作在林雁勤教授(通讯作者)的指导下完成,博士生罗尧为第一作者。共同作者还包括陈忠教授和屈小波教授。该项研究获得国家自然科学基金项目(22374124 和 22174118)的支持。
林雁勤教授团队近年来开展NMR谱图深度学习的研究,在谱图去噪、谱图重建以及纯化学位移谱图的获取方面都取得了进展(图4)。同时,林雁勤教授团队还全面总结分析了当前深度学习在NMR领域的应用(图4),让更多NMR领域的研究人员对深度学习有更深入的了解,推动NMR领域的进步。
论文链接:
https://www.nature.com/articles/s41467-025-57721-w
课题组网站:https://lingroup.xmu.edu.cn/
